Random forest といえば決定木を何本ももつアンサンブル学習で高い精度を持つ。 けど、どの説明変数が効いているかは説明が難しいものだと思いこんでいた。 ご近所のデータサイエンティストが R で効いている説明変数を出す方法を教えてくれたのでメモ。
ここでは R についてきている iris データセットを使う。 irisはアヤメの種類に関するデータセットで、1936年という大昔に フィッシャー が論文で使った歴史のあるデータセットでもある。 萼片 (sepal) の大きさ、花弁 (petal) の大きさ、アヤメの種類(species)が対になっている。
# iris dataset 読み込み
> data("iris")
> str(iris)
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
さて、random forest でアヤメを分類する。 手元にパッケージが入ってなかったのでそこから。
> install.packages("randomForest")
> library(randomForest)
> (fit <- randomForest(Species ~ ., data=iris))
Call:
randomForest(formula = Species ~ ., data = iris)
Type of random forest: classification
Number of trees: 500
No. of variables tried at each split: 2
OOB estimate of error rate: 4.67%
Confusion matrix:
setosa versicolor virginica class.error
setosa 50 0 0 0.00
versicolor 0 47 3 0.06
virginica 0 4 46 0.08
fit に random forest で学習したモデルを代入した。
重要な説明変数は importance() で取り出すことができる。
> importance(fit)
MeanDecreaseGini
Sepal.Length 10.499305
Sepal.Width 2.596317
Petal.Length 43.614641
Petal.Width 42.563816
どうやら花弁の大きさ(Petal.Length, Petal.Width)がアヤメの種類判定においては重要らしい。
varImpPlot(fit) すると importance(fit) と同じ結果をグラフにしてくれるようだ。
importance() では重要な説明変数が分かったが、目的変数に対してどう効くのか、この例でいえば Petal.Length が大きかったらどの種類になりやすいのか、分からない。
partialPlot() を使うと、どう影響するかが分かる。
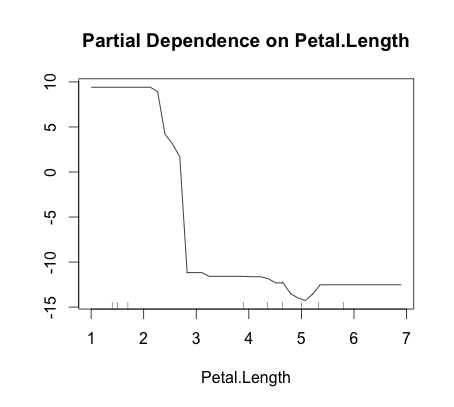
> partialPlot(fit, iris, Petal.Length, "setosa")
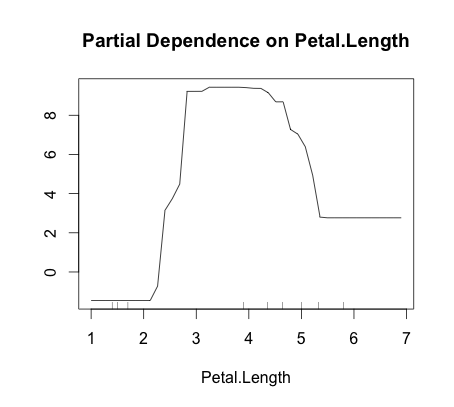
> partialPlot(fit, iris, Petal.Length, "versicolor")
> partialPlot(fit, iris, Petal.Length, "virginica")
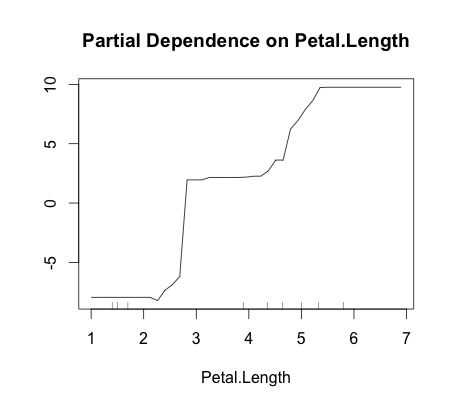
これを見るに、Petal.Length がおおよそ3より小さい時には setosa、 3から5の時には versicolor、5より大きい時には virginica である可能性が高いことが分かる。
まとめ
randomForest package の importance(), varImpPlot(), partialPlot() を使えば効いてる説明変数を見つけたり、効いている方向を確認したりできる。